生物と情報
小池 亮太郎(複雑系科学専攻)
生物とタンパク質とデータ
生物に関するいろいろな疑問を突き詰めていくと,なんだかんだでタンパク質に行きついたりする.“どうして私はお酒に弱いのか”ということを突き詰めると,体内にもっているアルコールを分解するタンパク質,アルコール脱水素酵素,の働きが弱いことが分かったり,“蛇に噛まれると何故まずいのか”を突き詰めると,蛇の唾液に含まれるタンパク質,ニューロトキシン,が毒で,そのタンパク質が人間の神経伝達を乱してしまうことが分かったりする.そして,神経伝達も複数のタンパク質の働きで成り立っていたりする.
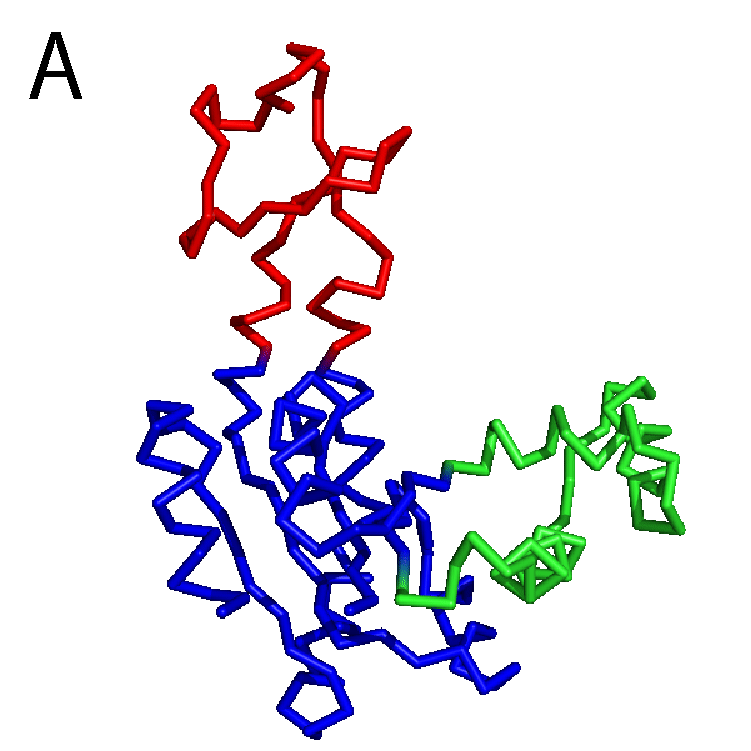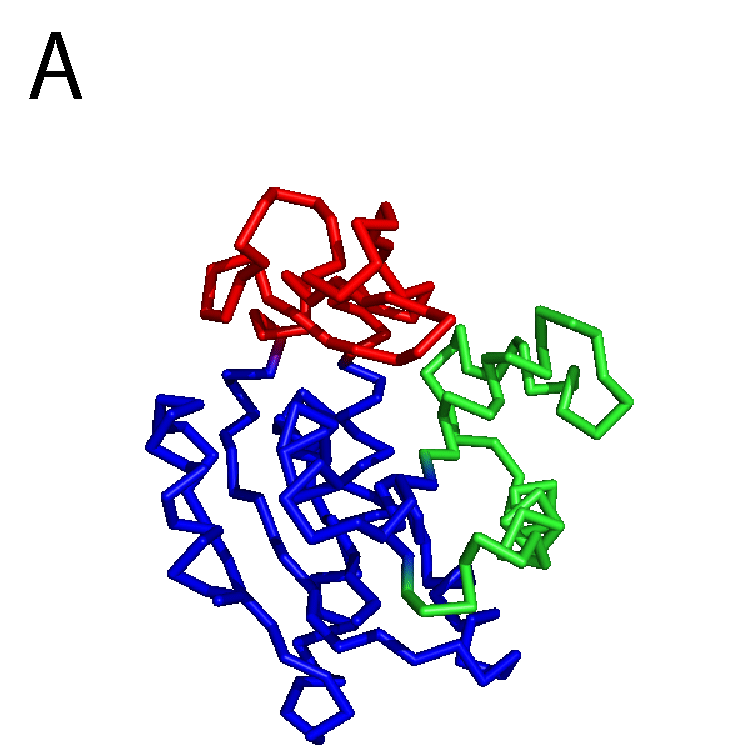
タンパク質はアミノ酸が数珠つなぎになったひも状の分子で,なかなかに複雑な「かたち」をしている.タンパク質の「かたち」のことを,これ以降は構造と呼ぶ.図1Aにアルコール脱水素酵素の構造を示す.なかなかに複雑,と言った気分を共有してもらえたなら幸いである.図1Bにはニューロトキシンの1種であるブンガロトキシンの構造を示す.アルコール脱水素酵素よりかはアミノ酸が少ないこともあり,やや簡単な構造に見えるだろうか.いずれにせよ,ひとくちにタンパク質といってもずいぶんと構造が異なっており,バラエティがあるのが分かる.
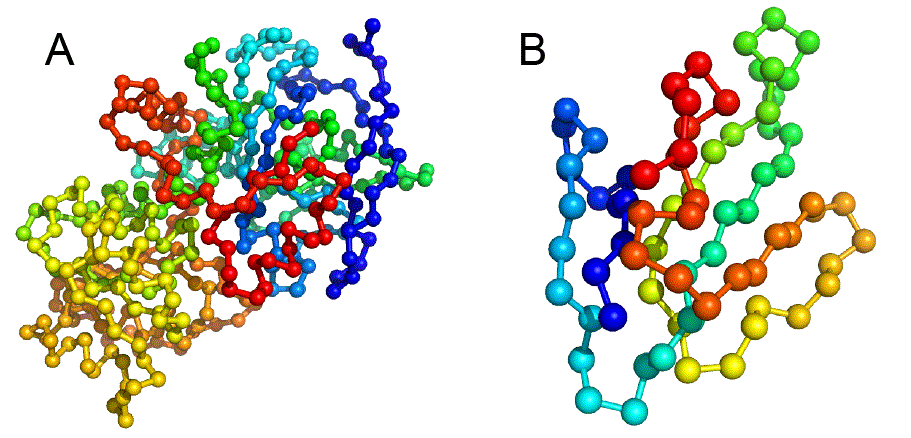
これらはほんのわずかな例に過ぎない.地球上にはたくさんの生物がいて,いろいろな働きをもっている.そういった働きは1つ,また1つと研究者らによって明らかにされてきた.その結果,膨大な数のタンパク質に関して,様々なことが明らかになっている.タンパク質に関する情報をもっとも広範にとりまとめたデータベースUniProt[1]では,2022年時点でおおよそ2億3千万ものタンパク質が登録されている.もちろん,まだまだ何をしているのかよく分かっていないタンパク質も大量にあり,きちんとレビューされたタンパク質は57万ほどである.また,構造が分かっているタンパク質も限られている.タンパク質の構造データはまた別のデータベースPDB(Protein Data Bank)[2]にまとめられており,2022年時点で20万ほどの構造データが登録されている.限られているとはいえ,20万という数字も十分に大きな値といえる.このように生物学の分野では,タンパク質に関する大量のデータが蓄積されており,現在も増大し続けている.そして,それらのデータはデータベースを通して公開されており誰でも使うことができる.
タンパク質の働きを可視化する
タンパク質に関するデータは,アミノ酸配列,構造,機能,タンパク質間の相互作用など多岐にわたるが,ここでは構造に関するデータをとりあげたい.タンパク質の構造のデータを利用することで,タンパク質が実際に働いている様子を見ることができる.先述したとおり,本当にたくさんのタンパク質がこの世界には存在するのだが,1つの例としてアデニル酸キナーゼというタンパク質を取り上げる.アデニル酸キナーゼは化学反応を触媒するタンパク質で,ATPとAMPからADPを2つつくる化学反応とその逆反応(ATP+AMP⇔2ADP)を触媒する.ATPは多くの生物で様々な仕事をするさいのエネルギー源として用いられる分子で,エネルギーを取り出すと,ADPへと変換される.多くのATPが消費され,ADPがたまってきても,アデニル酸キナーゼがあれば,ADPからATPへ戻すことができる.アデニル酸キナーゼはこの化学反応を触媒するために,ATPやAMP, ADPを認識し,特異的に結合する.AMPらを結合する前の構造と,結合した後の構造を並べることで,アデニル酸キナーゼがAMPらを結合する様子を見ることができる.結合前と後の構造は,先述したPDBから構造データを取得することで,パソコンやスマホで表示できる.ここでは,アデニル酸キナーゼがAMPとADPを結合する様子を動画(パラパラ漫画)にしたものを図2に示す.緑色のオブジェクトでアデニル酸キナーゼを,赤色のオブジェクトでADPを,オレンジ色のオブジェクトでAMP表示した.ステップ1ではアデニル酸キナーゼは何も結合しておらず,待ち受け状態にある.AMPらがやってくると(ステップ2),アデニル酸キナーゼはその構造を変化させ,AMPらをがっちりくわえこむ(ステップ3).こうしてアデニル酸キナーゼはくわえこんだAMPらに作用し,この化学反応を触媒する.このようにアデニル酸キナーゼの待ち受け状態(ステップ1)とADP,AMP結合状態(ステップ3)の構造を見比べることで,アデニル酸キナーゼがAMPらを結合する様子,すなわち働く現場,を見ることができる.
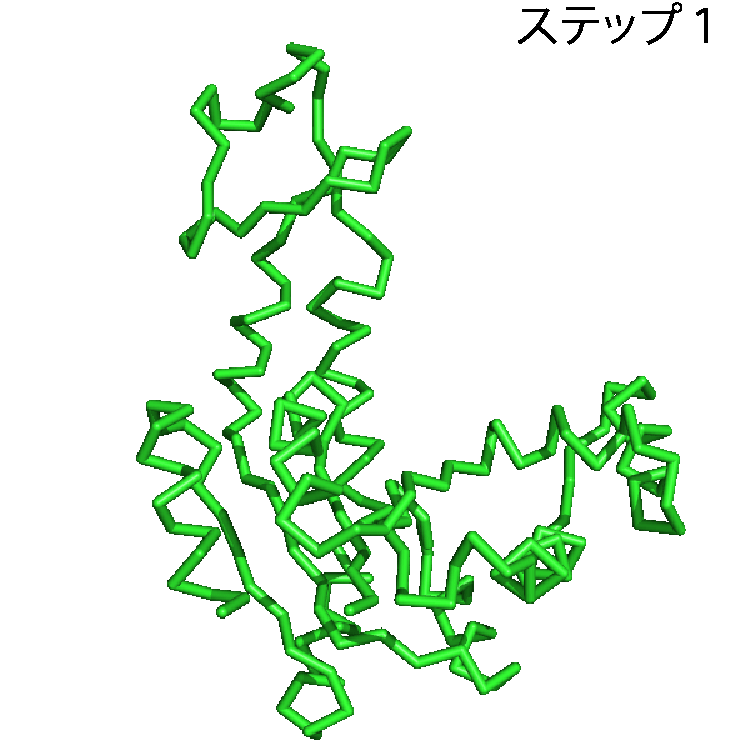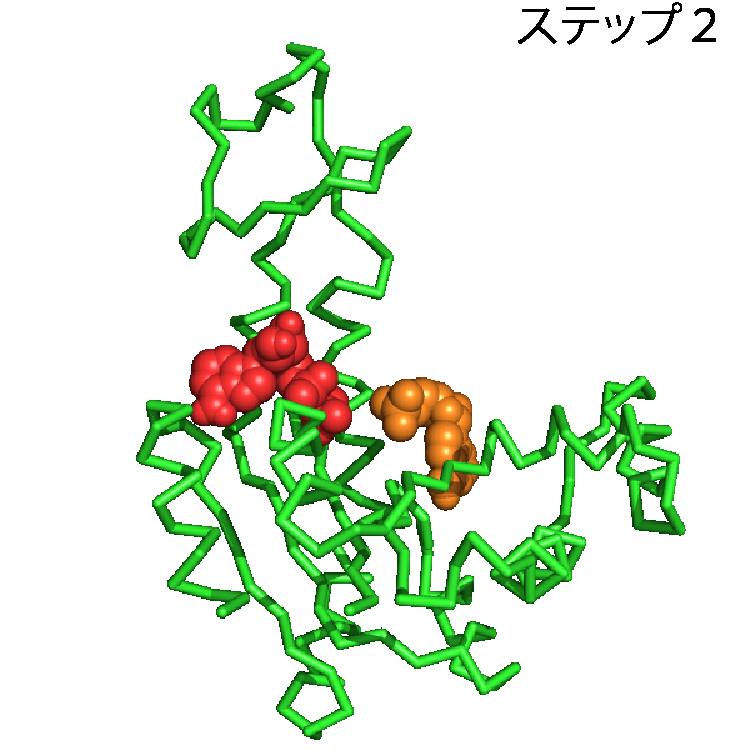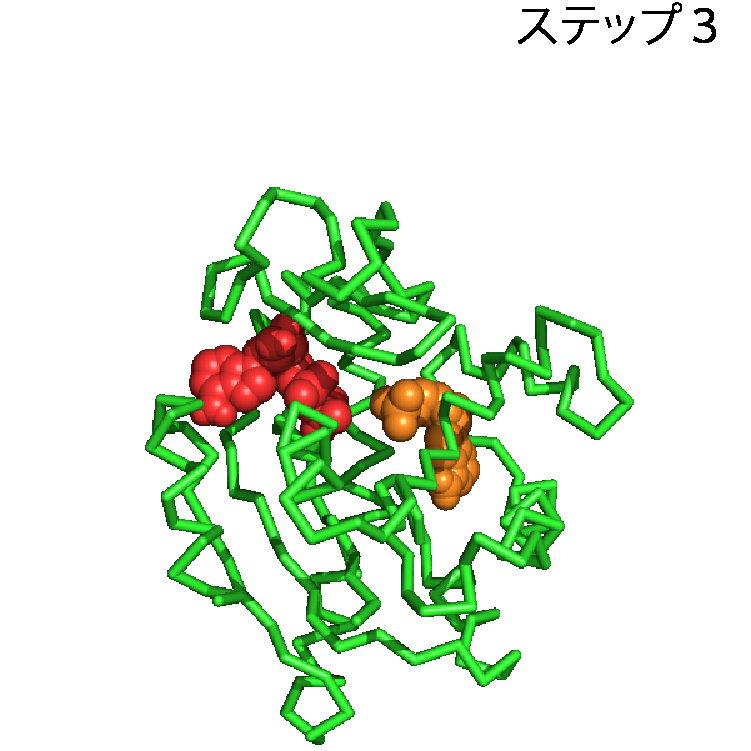
タンパク質の構造変化と剛体
アデニル酸キナーゼが働く,すなわちその機能を発揮するときに,その構造が変化するのは分かった.それでは,その構造変化とはどういったものなのだろうか?図2の動画を見せて「こんな様子です」と言うより,もっと詳細な解説や表現はできるだろうか?これに対する答えの1つは,そのタンパク質を構成する全ての原子あるいは全てのアミノ酸がどのように変化したかを列挙することである.例に示したアデニル酸キナーゼは214個のアミノ酸で構成されている.この場合,214個のアミノ酸がステップ1からステップ3の間にどこからどこまで動いたのかを記したリストを作れば良い.これはある意味とても正確な構造変化の表現ではあるが,214個ものアミノ酸の移動の情報をもらっても,そのままでは何が起こったか,人間には良くは分からないだろう.また,多くのタンパク質の構造変化では,全てのアミノ酸がてんでバラバラに動くのではなく,ある程度まとまって動くことが知られている.そのため,全てのアミノ酸の動きを列挙する代わりに,まとまって動くアミノ酸らをひとまとめにして,1つのかたまり(剛体)とみなし,少数の剛体の動きとして捉えた方が人間には分かりやすくなるだろう.
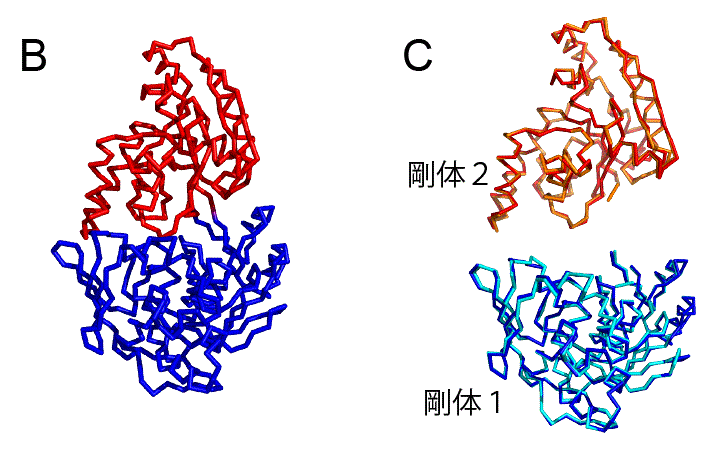
ここで剛体を特定するための考え方の1つを紹介する.あるタンパク質で構造が変化したとする.その様子を模式的に示したものを図3に示す.そのとき,同じ剛体を構成しているアミノ酸(図3のアミノ酸AとB)は,1つのかたまりとして同じように動くので,構造変化の前と後でその距離は変わらない.他方,別の剛体を構成するアミノ酸(図3のアミノ酸AとC,またはBとC)の距離は変化する.この性質を踏まえれば,構造変化の前後で距離が変わっていないアミノ酸ペアをグループにまとめていけば,剛体が特定できそうである.実際に,各アミノ酸ペアについて構造変化前と後での距離を測っておき,その差分を計算し,階層的クラスタリングの手法[3]を使って,差分が0に近いアミノ酸ペアからグループ化していくことで剛体を特定するソフト,Motion Tree,を筆者らは開発している[4].「剛体を特定するという問題」を,「距離の差分が0に近いものからグループ化する問題」と読み替えることで,既存のアルゴリズムである階層クラスタリングが適用可能な形式に,うまく持ち込んでいるわけである.
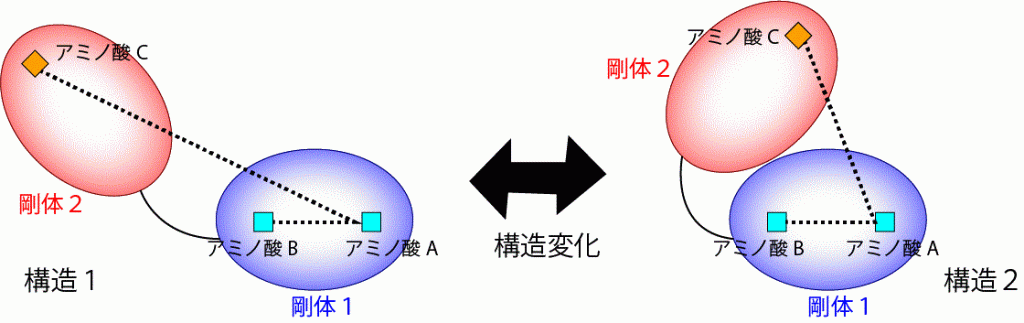
図3.タンパク質の構造変化と剛体の模式図.2つの剛体を青と赤の楕円で示した.また,剛体1の中のアミノ酸AとBを水色の四角で,剛体2の中のアミノ酸Cをオレンジ色の四角で表した.アミノ酸間の距離は点線で示した.
このソフトを先ほど示したアデニル酸キナーゼの構造変化に適用すると,複数の剛体が特定される.まずは3つの剛体が特定される(図4A).注目する対象は3つの剛体に絞られ,214個ものアミノ酸を追いかけずに済む.これにより,ずいぶんと簡単に構造変化の様子を把握することができる.ただ,実際のタンパク質の構造変化では完全な剛体になることはなく,剛体の中でも多少の変化が見られる.AMPらを結合する前と後で,各剛体でどんな構造変化が起こっているのか図4Bに示した.剛体1と剛体2では構造変化が小さく,結合前と後の構造が比較的よく重なっている.しかし,剛体3では構造変化が大きく,構造はあまり重ならない.そのため,この手法(Motion Tree)は剛体3をさらに分割し,2つの剛体を特定する(図4C).このようにアデニル酸キナーゼでは,タンパク質の全体の構造が大きく変わるようなグローバルな動き(図4Aの剛体2や3の動き)と,タンパク質の一部が変化するローカルな動き(図4Cの剛体3内部の構造変化)が見られることが分かった.一部の剛体の動きが階層的になっており,やや複雑ではあるが,214個のアミノ酸の動きを追うよりもずいぶんと簡単に構造変化を表現できる.
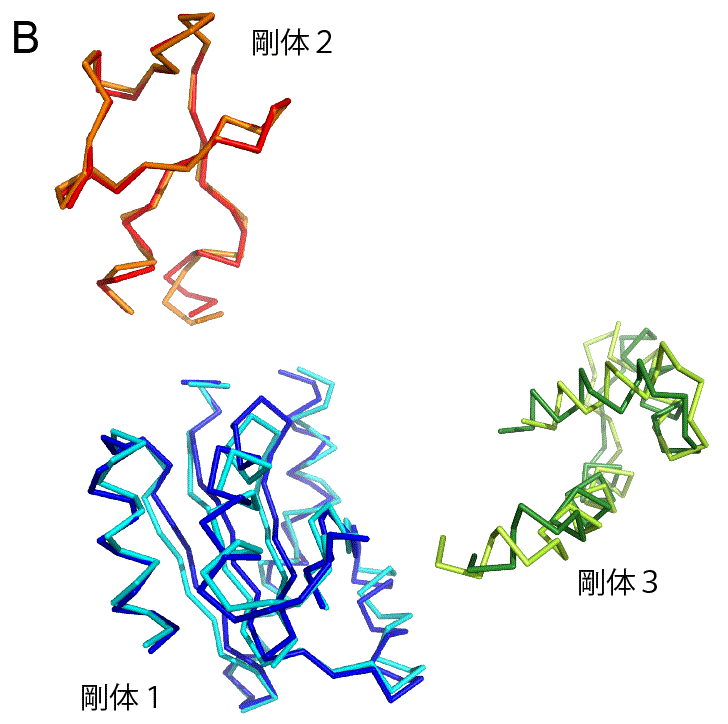
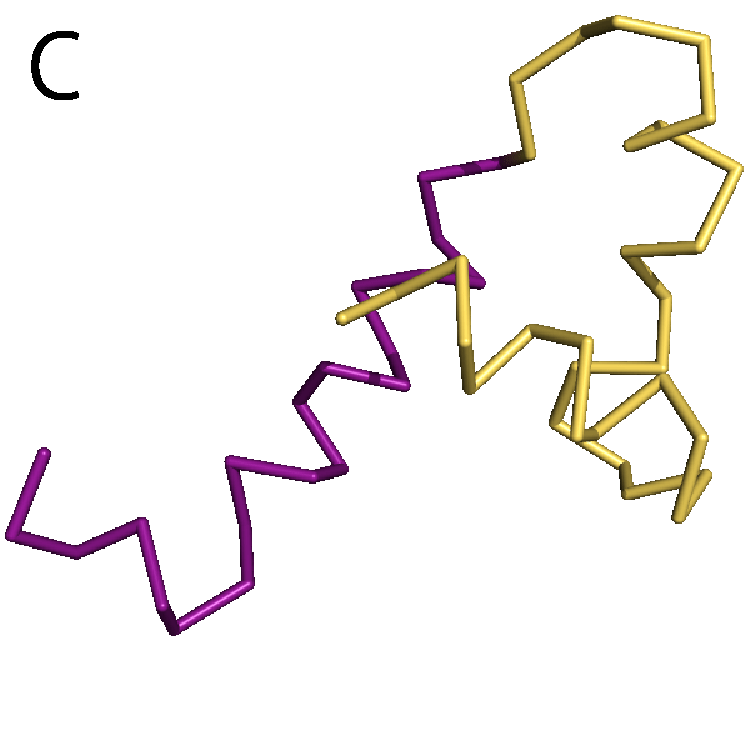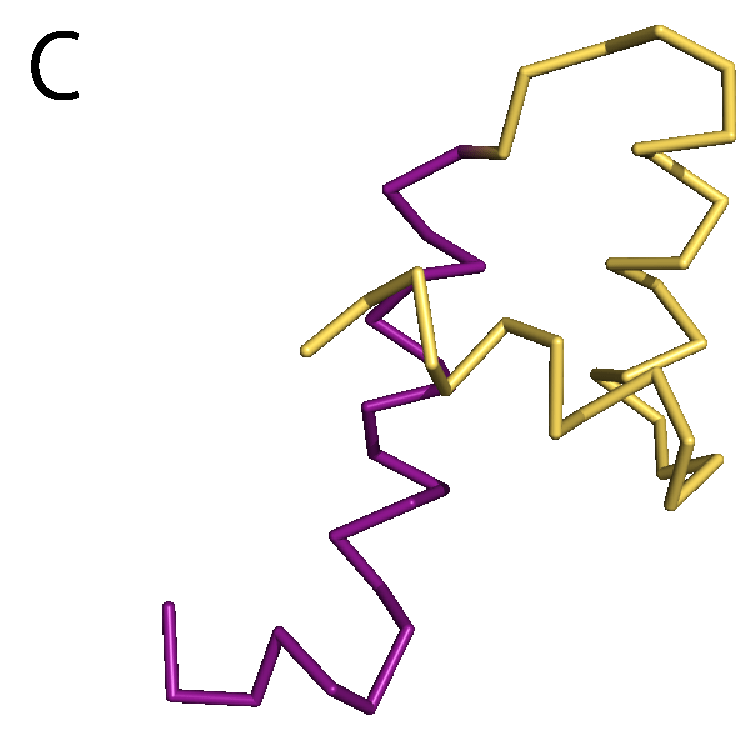
もう1つ別の構造変化の例として,DPBというタンパク質を取り上げる.DBPはペプチドと呼ばれるタンパク質の断片を運搬する機能をもっている.そのため,ペプチドを認識して,特異的に結合することができる.DPBが実際にペプチドを結合する様子を動画(パラパラ漫画)にしたものを図5Aに示す.緑色のオブジェクトでDPBを,赤色のオブジェクトでペプチドを表示した.ステップ1ではDPBはペプチドを結合しておらず,待ち受け状態にある.ペプチドがやってくると(ステップ2),DPBはその構造を変化させ,ペプチドを結合する(ステップ3).こうしてDPBは結合したペプチドを運搬していく.このDPBに,Motion Treeを適用すると,2つの剛体が特定される(図5B).これらの2つの剛体は構造変化前と変化後で,ほとんど構造が変わっておらず,完全な剛体に近い(図5C).また,DPBではアデニル酸キナーゼと違い,タンパク質全体の構造が変わるグローバルな運動は見られるが,ローカルな運動は見られなかった.タンパク質ごとに構造変化の様子も違うことが分かる.

大量のデータから知識を得る
専用のソフトを開発したのなら,大量にあるタンパク質の構造データに適用することができる.これによって,タンパク質全般では構造変化がどうなっているのか,調べることができる.ターゲットとなる分子(アデニル酸キナーゼの場合のAMPや,DPBのペプチドのこと)を結合する前と後というように,複数の構造データが利用可能なタンパク質を特定し,その中から代表的な424のタンパク質を選びだした.この424のタンパク質に先ほどのソフトMotion Treeを適用すると,アデニル酸キナーゼのように,グローバルな運動とローカルな運動の両方が見られるものが107,DPBのようにグローバルな運動しか見られないものが117,ローカルな運動しかみられないものが177,例外的なものが23,となることが分かった[4].また,これらのタンパク質の働きを調べてみると,両方の運動が見られるグループではメチル基やリン酸基などを転移させる反応を触媒するタンパク質が,グローバルな運動が見られるグループではDNAを結合するものが,ローカルな運動が見られるグループでは加水分解反応を触媒するタンパク質が,それぞれ比較的多くみられることが分かった.どのような構造変化をするかによって,タンパク質の働きが異なる様子がうかがえる.このように専用のソフトを大量のデータに適用することで,ぼんやりとではあるがタンパク質全体の様子を探ることもできる.
おわりに
ものごとを分かりやすく見たい,というモチベーションがある.複雑な対象でも,できるだけその複雑さがもつエッセンスは損なわずに,それでいてできるだけシンプルな見方ができるとなお良い.タンパク質の構造変化を見るときでも,やはり分かりやすく見たいと思い,剛体を特定するソフトを開発した.剛体を特定し,そこの色を変えるだけでも,複雑な構造変化がだいぶ分かりやすく見えるようになったと思う.ただ,タンパク質の周辺にはまだまだ複雑な様相のものも多い.そういったものを分かりやすく見るための,見方や枠組み,アルゴリズムなどを今後も開発していければな,と考えている.
最後に,この文章が読者の方にそれなりに分かりやすいものになっていれば幸いである.そうでないと感じた方が,分かりやすく改変してくれるソフトを開発してくれるならばさらに幸いである.
参考文献
[1] The UniProt Consortium, UniProt: the universal protein knowledgebase in 2021, Nucleic Acids Res, 49(D1), D480-D490 (2021). https://doi.org/10.1093/nar/gkaa1100
[2] wwPDB consortium, Protein Data Bank: the single global archive for 3D macromolecular structure data, Nucleic Acids Res, 47(D1), D520-D528 (2019). https://doi.org/10.1007/978-1-4939-7000-1_26
[3] 宮本定明,クラスター分析入門,森北出版(1999).
[4] R Koike, M Ota and A Kidera, Hierarchical description and extensive classification of protein structural changes by Motion Tree, J Mol Biol, 426, 752-762 (2014). https://doi.org/10.1016/j.jmb.2013.10.034
[5] BJ Gibbons and TD Hurley, Structure of three class I human alcohol dehydrogenases complexed with isoenzyme specific formamide inhibitors, Biochemistry, 43, 12555-12562 (2004). https://doi.org/10.1021/bi0489107
[6] M Harel et al, The binding site of acetylcholine receptor as visualized in the X-Ray structure of a complex between alpha-bungarotoxin and a mimotope peptide, Neuron, 32, 265-275 (2001). https://doi.org/10.1016/s0896-6273(01)00461-5
[7] CW Müller, GJ Schlauderer, J Reinstein and GE Schulz, Adenylate kinase motions during catalysis: an energetic counterweight balancing substrate binding, Structure, 4, 147-156 (1996). https://doi.org/10.1016/s0969-2126(96)00018-4
[8] MB Berry, E Bae, TR Bilderback, M Glaser and GN Phillips Jr, Crystal structure of ADP/AMP complex of Escherichia coli adenylate kinase, Proteins, 62, 555-556 (2006). https://doi.org/10.1002/prot.20699
[9] AV Nickitenko, S Trakhanov and FA Quiocho, 2 Å resolution structure of DppA, a periplasmic dipeptide transport/chemosensory receptor, Biochemistry, 34, 16585-16595 (1995). https://doi.org/10.1021/bi00051a006
[10] P Dunten and SL Mowbray, Crystal structure of the dipeptide binding protein from Escherichia coli involved in active transport and chemotaxis, Protein Sci, 4, 2327-2334 (1995). https://doi.org/10.1002/pro.5560041110

