特集「新型コロナと情報」:新型コロナのPCR法
今日は分子生物学が専門の青木摂之さん(実験する人:以下A)にバイオインフォマティクスを専門とする太田元規(実験しない人:以下O)が質問する形で,新型コロナの検査に用いられるPCR法について紹介したいと思います.
O: まず断っておきたいのは,この企画は一般的なPCR法の説明をするものではありません.「新型コロナ」のPCRなので,そこに限定して突っ込みを入れたいと思います.一般的なPCR法の解説はどこか他のサイトを見てくださいね.では早速ですが,まず新型コロナのゲノムはRNAですよね.PCRはDNAを増やす方法だから,PCRをやるだけじゃダメですよね.
A: まず,RNAを鋳型にしてDNAを合成する逆転写酵素という酵素を使って,新型コロナのゲノムRNAに対応するDNA(相補的DNA, cDNA)を作ります.いわゆる逆転写(RT)反応で,PCRとあわせてRT-PCRと言っています.
O: この段階では増殖させるの?cDNAはちょっとあればいいの?
A: ちょっとあればいいです.あとでPCRで増やすので.
O: PCRでは好熱菌(高温で生きている菌)の酵素を使うんだけど,逆転写酵素は普通の酵素でもいいの?
A: 普通でいいです.
O: 逆転写反応もプライマー必要ですよね.
A: プライマー,使います.ポリTプライマーとか,ランダムプライマーとか.感染症研(国立感染症研究所)が発表しているRT-PCRのプロトコル(工程マニュアル)では,両方混ぜて使ってますね。
O: プライマーって特異性なくてもいいの?
A: 逆転写のときは,かならずしも特異的なプライマーじゃなくてもいいです.後でPCRで特異性を出せるわけです.
O: じゃあこの時はヒトのメッセンジャーRNAとか,なんでもcDNAになっちゃうのね.
A: ただこの場合は,感染してヒトの細胞の中にいるウイルスのRNAを抽出するんじゃなくて、すでに細胞から出てきたウイルスを含む「鼻咽頭ぬぐい液」(とか最近だと唾液でもいいことになったみたいです)からRNAを抽出するので,いろんな配列が混ざってしまう心配は要らないようですね.
O: それで,じゃあ次にDNAを増やしましょう,ということでPCRをやるんだけど,逆転写反応とPCRは同じ装置でできるの?
A: 感染症研のリアルタイムPCRのプロトコルでは,そのままでやってしまう方法をとってます.最初の30分くらいは温度を50℃にして逆転写反応を進めて,その後,同じチューブの中でPCRのプロセスに入ります.まあこうした反応自体は結局市販のキットを使ってるわけですが.
O: なるほど.流れ作業でいけるのね.では次にPCRをやるのだけど,この時にはあるDNAを狙って増殖させるわけですよね.その遺伝子って何なの?
A: 今公開されている全部の新型コロナ検査キットは知らないのだけど...
O: 検査キットは一杯出てるの?
A: 結構出てるようですが,感染症研が出している標準プロトコルに従っているものが多いと思うから,すくなくとも基本的なところは,そんなに多様性はないんじゃないかな?感染症研では,リアルタイムPCRと普通のPCR(増幅の結果を電気泳動で検出する従来の方法)の2つのプロトコルを出してます.
O: リアルタイムPCRってなんか特別なヤツですか?
A: DNAの検出方法が違うんです.電気泳動はアガロースゲルに電圧かけてDNAを陰極側から陽極側へ移動させます.短いDNAの方がゲル内部の網目状の穴を早く移動できるので、移動の距離を見ることでDNAのサイズを推定できるわけです。感染症研のプライマーの場合だと,200〜300 bpくらいの長さのバンドが出るかを見ます.
O: 分子量みたいなものを見ているんでしょ.でもそれだとDNA配列の特異性が出ないよね.
A: 特異性は出なくていいんです.それはプライマーで見るから.
O: 要するに,PCRが起きているかどうかのチェックをするだけなんだ.
A: でも一応感染症研では,最初にPCRする時にはシーケンシング(塩基配列の決定)をして新型コロナウイルスかどうかを確認してね,と言ってます.
O: わかりました.リアルタイムの方は?
A: PCRが起こると増幅した分だけ蛍光が発するような工夫をしておいて,反応させるのと同時に検出するので,感度が良く,定量性も高いのです.
O: ほう.すごそうですね.それで,プライマーは?
A: 従来型のPCRだと,ゲノムRNAを複製したりする酵素など,複数のタンパク質の情報をコードしている1a遺伝子と,スパイクタンパク質をコードするS遺伝子の2つがターゲットですね.
O: うん,そうなんだけど,新型コロナって旧型コロナ,つまり風邪のウイルスの仲間だから,間違って旧型の遺伝子を増やしちゃうと,ただの風邪でも新型として判定されちゃう.だから,結構他の近縁種とは配列が違う遺伝子をターゲットにしないといけない.その点,それらのターゲット配列はどうなの?
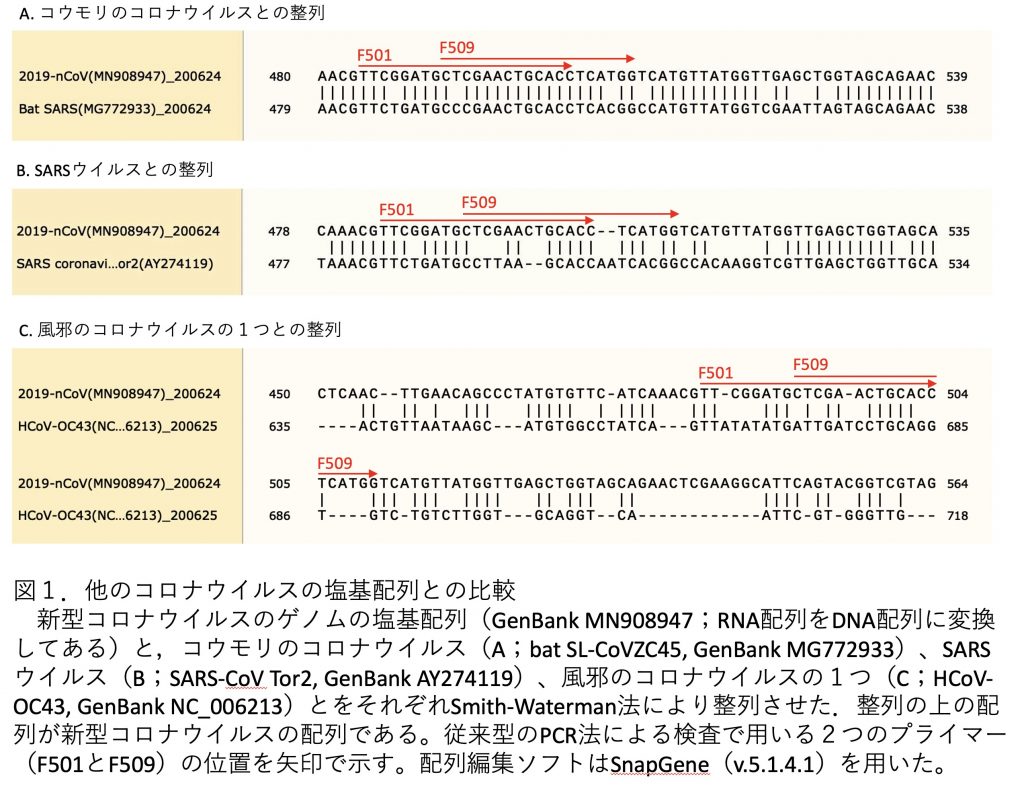
A: 下の図1をみてください.新型コロナと一番似ているウイルスはコウモリのコロナウイルスの一種で,配列一致度は90%くらい(図1A).よく似てますね.まあ似てるからこそ新型コロナはもともとコウモリを宿主としてたコロナが起源だ,という話になってる訳ですが.これだけ似てると,コウモリコロナを新型コロナと間違えて判定しちゃう可能性はありますね.だけど,コウモリのコロナを新型コロナと間違える心配は現実にはほとんどないとは思いますが.
O: なるほど.でもさ,風邪コロナだとどうですか.
A: いわゆる風邪のコロナは4種類あって,その中の2つは,新型コロナや,SARSやMERSを引き起こすコロナと同じβコロナウイルスって言うんですが,これだと配列一致度は50%ちょっと(図1C).
O: 50%ちょっとですか.結構違うね.
A: うんうん.これなら,経験的なことしか言えないんだけど,おそらく間違えません.
O: でもプライマーのところ,似てません?
A: プライマーは5’末端側は似ていてもそんなに問題ないです.3’末端が合わないと次の合成ができないから,3’末端側さえちゃんと違っていればだいたい大丈夫です.
O: ほう.それは知らなかった.
A: あと感染症研のやり方だとNested PCRというのを推奨していて,2段階でPCRをやるんです.これはプライマーを2セット使ってPCRします.1回目で増やす領域と2回目で増やす領域を入れ子にする.こうすると2回ともPCRに引っかからないと増えないので,確実性が増します.
O: なるほど.
A: だから,風邪のコロナは間違えないので大丈夫です.MERSも結構配列が違うので間違えません.でもSARSは配列一致度が80%ほどもあるので(図1B),これはプライマーの配列や実験の条件によっては間違えるかもしれない.だけど,日本ではSARSは流行ってないし,さしあたり問題ないと思います.
O: うんうん.PCRで,新型コロナをちゃんと他のコロナと区別して判定できそうなのは分かりました.でもどうしてここの配列なのかな?RNAゲノムの他の配列じゃダメなの?
A: PCRのプライマーに向いてますよって配列があるんですよね.例えばGCだとATに比べて水素結合を多く作るので,GCが多すぎるとベタベタくっついちゃって,非特異的な結合をする場合がある.そうするとPCRが始まっちゃう場合があるんです.繰り返し配列もダメです.特異性が低いので.ATGCそれぞれ適当に入っていてGCが多すぎないのがいいんです.どこでもプライマーに向いているわけじゃない.
O: じゃあ全部のキットで1aとSをターゲット配列として使っているんですか?
A: 1aとSは従来型のPCRに基づく方法のターゲットで,リアルタイムPCRによる方法では,ヌクレオキャプシドというタンパク質をコードするN遺伝子の配列を使ってるようです.少し調べたところでは,市販のキットでも同じプライマーを使ってますね.検出時間が短くてすんだり,いろいろ変法はあるようですが,基本は感染症研のプロトコルに準じたやり方になっているんだと思います.
O: リアルタイムPCRで利用する蛍光性のDNA配列はどのヘンになるんですか?
A: プライマーで利用するところの真ん中ヘンに入れておきますね.
O: なるほど.完璧ですね.これでだいたいちょっと疑問に思っていたことは解決しました.こういう方法なら風邪のコロナとの識別もできますね.よくわかりました.青木さん,どうもありがとうございました.
A: どういたしまして!

